您现在的位置: 首页» 学院动态» 【科研新进展】姜雨教授团队在山羊泛基因组和结构变异研究方面取得重要进展
【科研新进展】姜雨教授团队在山羊泛基因组和结构变异研究方面取得重要进展
作者:巩敏芝 发布日期:2024-12-13 浏览次数:
12月12日,姜雨教授团队在国际期刊Molecular Biology and Evolution上发表了题为《A graph-based goat pangenome reveals structural variations involved in domestication and adaptation》的研究论文,构建了山羊的图结构泛基因组和结构变异集合。研究生边培培、李嘉鑫和周士硕为该论文第一作者,我校姜雨教授和王喜宏副教授为该论文的通讯作者。

图1 山羊基因组结构变异检测流程
泛基因组技术具备捕捉一个物种内部所有遗传多样性的能力,为我们提供了一个全面的遗传变异视角。在这些变异中,结构变异(≥50 bp)相较于单核苷酸多态性和小的插入缺失,在基因和调控区域引起的基因组变化更为显著,对个体的表型多样性有着重要影响,并与生长、适应性和疾病易感性等特征紧密相关。本项目为8个具有代表性的山羊品种组装了高质量的单倍型基因组,并构建了图结构的泛基因组,用于结构变异的检测和分型。此外,我们还利用20个山羊样本的PacBio HiFi低深度测序数据,为结构变异的检测提供了进一步的支持,总计获得了超过17万个结构变异和113Mb的新序列。
本团队在之前的工作中已经鉴定出了两个关键的驯化基因抗寄生虫的MUC6座位和警惕行为改变的RRM1座位。这次利用山羊泛基因组,对结构变异在1227只野山羊和家山羊进行了基因分型比较,揭示了一个新的抗病驯化基因座位。该座位通过基因复制产生四个新的galectin-9 like基因( LGALS9L ),其突变频率约在4000年前显著上升。单细胞数据显示这些新复制基因的表达具有高度的组织和细胞特异性,暗示了基因复制以后的功能分化。
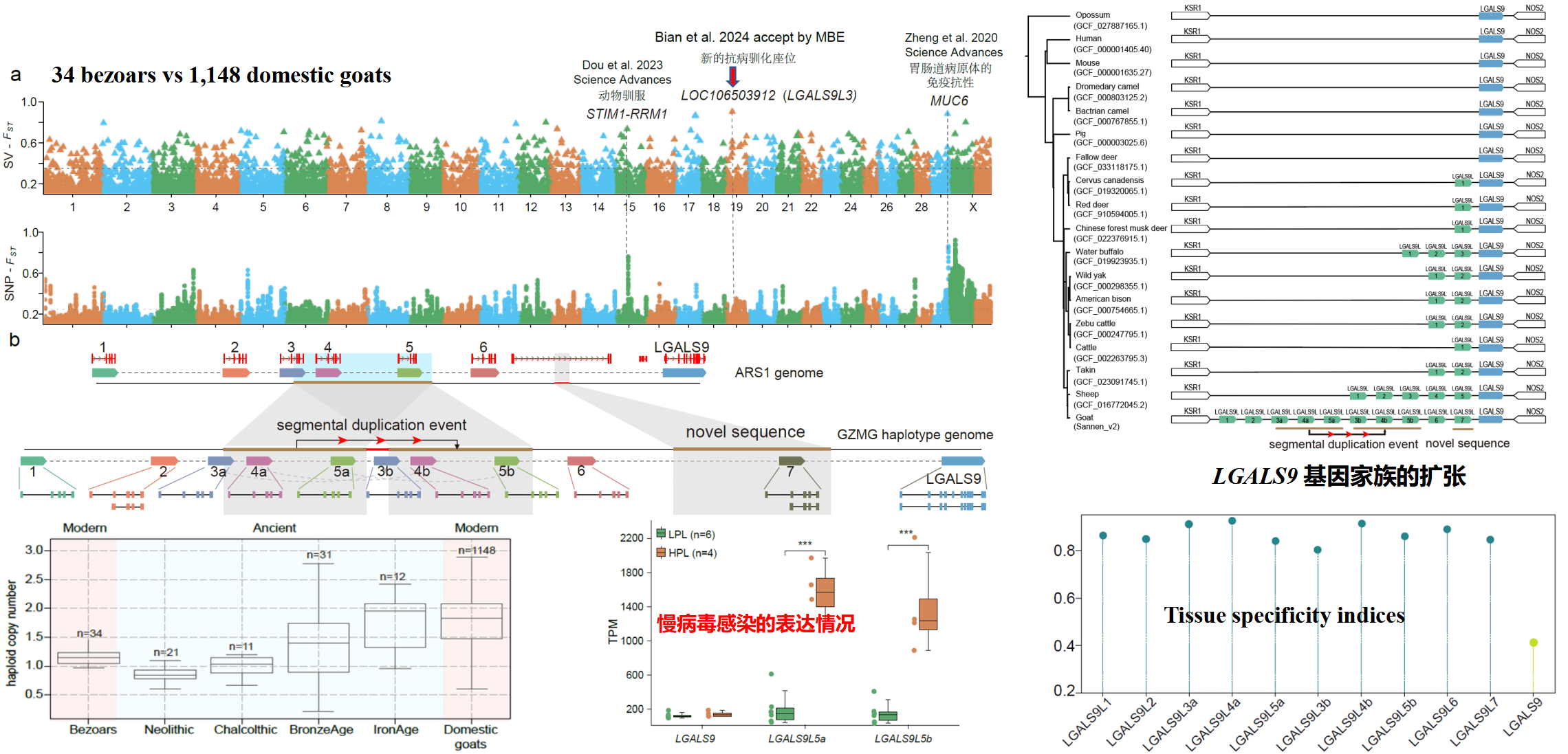
图2 驯化过程中 LGALS9L 基因拷贝的增加
本研究得到国家重点研发计划(2022YFF1000100)、陕西省畜禽育种双链融合重点项目(2022GD-TSLD-46-0401)、新疆维吾尔自治区科技重大专项(2024A02004)等项目的资助。感谢西北农林科技大学高性能计算平台提供计算资源以及榆林市羊产业研究院提供样本采集。
原文链接:
编辑:巩敏芝 终审:赵运良
